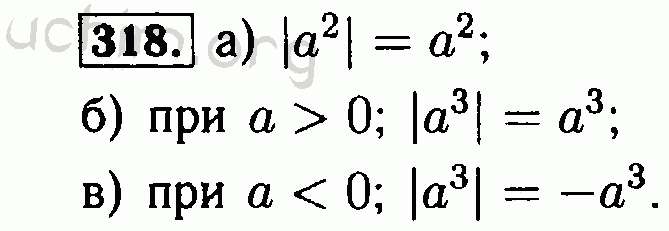Помогите определить сторону прямоугольника 318 Алгебра 9 класс Макарычев – Рамблер/класс
Помогите определить сторону прямоугольника 318 Алгебра 9 класс Макарычев – Рамблер/классИнтересные вопросы
Школа
Подскажите, как бороться с грубым отношением одноклассников к моему ребенку?
Новости
Поделитесь, сколько вы потратили на подготовку ребенка к учебному году?
Школа
Объясните, это правда, что родители теперь будут информироваться о снижении успеваемости в школе?
Школа
Когда в 2018 году намечено проведение основного периода ЕГЭ?
Новости
Будет ли как-то улучшаться система проверки и организации итоговых сочинений?
Вузы
Подскажите, почему закрыли прием в Московский институт телевидения и радиовещания «Останкино»?
Одна сторона прямоугольника на 7 см больше другой. Какой
Какой
не превосходит 60 см2?
ответы
Приветик. Помогу
ваш ответ
Можно ввести 4000 cимволов
отправить
дежурный
Нажимая кнопку «отправить», вы принимаете условия пользовательского соглашения
похожие темы
ЕГЭ
10 класс
11 класс
Химия
похожие вопросы 5
150 Алгебра 9 класс Макарычев Помогите решить графически
Решите графически уравнение:
а) х3 = 2; б) х3 = 4; в) х3 = -5.
ЭкзаменыАлгебра9 классМакарычев Ю.Н.ГДЗ
Когда скорость изменения функции будет наибольшей или наименьшей? Алгебра 10-11 класс Колмогоров Упр 308
Совсем я в точных науках не сильна) Кто поможет?) Найдите значения аргумента из промежутка [-2; 5], при которых скорость изменения (Подробнее.
ГДЗ11 классКолмогоров А.Н.Алгебра
Васильевых. 50 вариантов ответов по русскому языку. Вариант 31 ч.2 Задание 13 ОГЭ Русский язык 9 класс Однородное подчинение придаточных
Среди предложений 21-29:
(21) И Митрофанов услышал в этом смехе и прощение себе, и даже какое-то (Подробнее…)
ГДЗРусский языкОГЭ9 классВасильевых И.П.
16. Расставьте все знаки препинания: укажите цифру(-ы), на месте которой(-ых)… Цыбулько И. П. Русский язык ЕГЭ-2017 ГДЗ. Вариант 13.
16.
Расставьте все знаки препинания: укажите цифру(-ы), на месте которой(-ых)
в предложении должна(-ы) стоять запятая(-ые). (Подробнее…)
ГДЗЕГЭРусский языкЦыбулько И.П.
ЕГЭ-2017 Цыбулько И. П. Русский язык ГДЗ. Вариант 13. 18. Расставьте все знаки препинания: укажите цифру(-ы), на месте которой(-ых)…
18.
Расставьте все знаки препинания: укажите цифру(-ы), на месте которой(-ых)
в предложении должна(-ы) стоять запятая(-ые). (Подробнее…)
(Подробнее…)
ГДЗЕГЭРусский языкЦыбулько И.П.
Быстрая и экономичная по памяти scRNA-seq кластеризация k-средних с различными расстояниями
[1] Ahn Euijoon, Kumar Ashnil, Feng Dagan, Fulham Michael J. и Kim Jinman. 2019. Неконтролируемое изучение признаков с помощью K-средних и ансамбля глубоких сверточных нейронных сетей для классификации медицинских изображений. CoRR, архив: 1906.03359 (2019). arXiv:1906.03359 [Google Scholar]
[2] Артур Давид и Васильвицкий Сергей. 2007. K-Means++: преимущества тщательного посева. СОДА (2007), 1027–1035. [Академия Google]
[3] Бейкер Дэниел. 2008. libsimdsampling. http://github.com/dnbaker/libsimdsampling. [В сети; по состоянию на 7 февраля 2021 г.].
[4] Балкан Мария-Флорина Ф., Эрлих Стивен и Лян Иньюй. 2013. Распределенная k -средняя и k -медианная кластеризация на общих топологиях. Достижения в области нейронных систем обработки информации
26 (2013), 1995–2003 гг. [Google Scholar]
[Google Scholar]
[5] Банерджи Ариндам, Меругу Сруджана, Диллон Индерджит С. и Гош Джойдип. 2005. Кластеризация с расхождениями Брегмана. Журнал исследований машинного обучения 6, 58 (2005), 1705–1749.. http://jmlr.org/papers/v6/banerjee05b.html [Google Scholar]
[6] Боту Леон и Бенжио Йошуа. 1995. Свойства сходимости алгоритмов k-средних. В Достижениях в области нейронных систем обработки информации. 585–592. [Google Scholar]
[7] Brennecke P, Anders S, Kim JK, Kolodziejczyk AA, Zhang X, Proserpio V, Baying B, Benes V, Teichmann SA, Marioni JC и Heisler MG. 2013. Учет технического шума в экспериментах по секвенированию РНК на одной клетке. Нат Методы 10, 11 (ноябрь 2013), 1093–1095. [PubMed] [Google Scholar]
[8] Бродер Андрей, Гарсия-Пуэо Луис, Иосифовский Ваня, Васильвицкий Сергей и Венкатесан Шрихари. 2014. Масштабируемые K-средние с помощью ранжированного поиска. В материалах 7-й Международной конференции ACM по поиску в Интернете и интеллектуальному анализу данных
(Нью-Йорк, Нью-Йорк, США) ( WSDM ’14 ). Ассоциация вычислительной техники, Нью-Йорк, штат Нью-Йорк, США, 233–242. 10.1145/2556195.2556260 [CrossRef] [Google Scholar]
Ассоциация вычислительной техники, Нью-Йорк, штат Нью-Йорк, США, 233–242. 10.1145/2556195.2556260 [CrossRef] [Google Scholar]
[9] Cao J, O’Day DR, Pliner HA, Kingsley PD, Deng M, Daza RM, Zager MA, Aldinger KA, Blecher-Gonen R, Zhang F, Spielmann M, Palis J, Doherty D, Steemers FJ, Glass IA, Trapnell C, and Shendure J. 2020. Атлас клеток человека по экспрессии генов плода. Наука 370, 6518 (ноябрь 2020). [Бесплатная статья PMC] [PubMed] [Google Scholar]
[10] Цао Дж., Шпильманн М., Цю Х., Хуан Х., Ибрагим Д.М., Хилл А.Дж., Чжан Ф., Мундлос С., Кристиансен Л., Стимерс Ф.Дж., Трапнелл С. и Шендур Дж. 2019. Одноклеточный транскрипционный ландшафт органогенеза млекопитающих. Природа 566, 7745 (02 2019), 496–502. [Бесплатная статья PMC] [PubMed] [Google Scholar]
[11] Chen Xinlei и Cai Deng. 2011. Крупномасштабная спектральная кластеризация с представлением на основе ориентиров. В материалах двадцать пятой конференции AAAI по искусственному интеллекту
(Сан-Франциско, Калифорния) ( AAAI’11 ). AAAI Press, 313–318. [Google Scholar]
AAAI Press, 313–318. [Google Scholar]
[12] Якоб Венцель Лемир Даниэль. 2013. СИМДПКГ. https://github.com/lemire/simdpcg.
[13] Датлингер Пол, Рендейро Андре Ф, Бенке Торина, Краусгрубер Томас, Баррека Даниэле и Бок Кристоф. 2019. Секвенирование одноклеточной РНК со сверхвысокой пропускной способностью с помощью комбинаторной жидкостной индексации. биоРксив (2019). https://doi.org/10.1101/2019.12.17.879304 arXiv: https://doi.org/10.1101/2019.12.17.879304https://www.biorxiv.org/content/early/2019/12/18/2019.12.17.879304.full.pdf arXiv: https://www.biorxiv.org/content/early/2019/12/18/2019.12.17.879304.full.pdf
[14] ДеМео Б. и Бергер Б. 2020. Хоппер: математически оптимальный алгоритм зарисовки биологических данных. Биоинформатика 36 (07 2020), i236–i241. [Бесплатная статья PMC] [PubMed] [Google Scholar]
[15] Элкан Чарльз. 2003. Использование неравенства треугольника для ускорения K-средних. В материалах двадцатой международной конференции по машинному обучению
(Вашингтон, округ Колумбия, США) ( ICML’03 ). АААИ Пресс, 147–153. [Google Scholar]
АААИ Пресс, 147–153. [Google Scholar]
[16] Фельдман Дэн и Лангберг Майкл. 2011. Единая структура для аппроксимации и кластеризации данных. CoRR абс/1106.1379 (2011). arXiv:1106.1379 http://arxiv.org/abs/1106.1379
[17] Хикс Стефани К., Лю Руокси, Ни Ювэй, Пурдом Элизабет и Риссо Давиде. 2021. mbkmeans: быстрая кластеризация данных по одной ячейке с использованием мини-пакетных k-средних. PLOS Вычислительная биология 17, 1 (января 2021), 1–18. 10.1371/journal.pcbi.1008625 [бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
[18] Хи Б., Чо Х., ДеМео Б., Брайсон Б. и Бергер Б. 2019. Геометрические наброски компактно обобщают транскриптомный ландшафт одноклеточных. Сотовая система 8, 6 (июнь 2019), 483–493. [Бесплатная статья PMC] [PubMed] [Google Scholar]
[19] Hübschle-Schneider Lorenz and Sanders Peter. 2020. Коммуникационный (взвешенный) отбор проб коллектора из полностью распределенных потоков данных. КОРР (2020). arXiv:1910.11069 [cs.DS] [Google Scholar]
[20] Латтанци Сильвио и Солер Кристиан. 2019. Улучшенный алгоритм k-means++ с помощью локального поиска. В материалах 36-й Международной конференции по машинному обучению (Материалы исследований в области машинного обучения, том 97), Чаудхури Камалика и Салахутдинов Руслан (ред.). PMLR, 3662–3671. http://proceedings.mlr.press/v97/lattanzi19a.html [Google Scholar]
2019. Улучшенный алгоритм k-means++ с помощью локального поиска. В материалах 36-й Международной конференции по машинному обучению (Материалы исследований в области машинного обучения, том 97), Чаудхури Камалика и Салахутдинов Руслан (ред.). PMLR, 3662–3671. http://proceedings.mlr.press/v97/lattanzi19a.html [Google Scholar]
[21] Лемир Даниэль. 2016-2018 гг. SIMDPCG. https://lemire.me/blog/2018/06/07/vectorizing-random-number-generators-for-greater-speed-pcg-and-xorshift128-avx-512-edition/. [Академия Google]
[22] Ли Цюхун, Ван Пэн, Ван Вэй, Ху Хао, Ли Чжуншэн и Ли Цзюньсянь. 2014. Эффективный алгоритм кластеризации K-средних на MapReduce. В системах баз данных для продвинутых приложений Бхоумик Сурав С., Дайресон Кертис Э., Дженсен Кристиан С., Ли Монг Ли, Мулиантара Агус и Тальхейм Бернхард (ред.). Springer International Publishing, Чам, 357–371. [Google Scholar]
[23] Ллойд Стюарт П.. 1982.
Квантование методом наименьших квадратов в pcm. IEEE Transactions по теории информации
28 (1982), 129–137.
[24] Ллойд Стюарт П.. 1982. Квантование методом наименьших квадратов в PCM. IEEE транс. Теория информации 28 (1982), 129–136. [Google Scholar]
[25] Люцик Марио, Бахем Оливье и Краузе Андреас. 2016. Сильные базовые наборы для жесткой и мягкой кластеризации Брегмана с приложениями к смесям экспоненциальных семейств. КОРР (2016). arXiv:1508.05243 [stat.ML] [Google Scholar]
[26] Макарычев Константин, Макарычев Юрий, Разенштейн Илья П.. 2018. Производительность преобразования Джонсона-Линденштрауса для кластеризации k-средних и k-медиан. CoRR абс/1811.03195 (2018). arXiv:1811.03195 http://arxiv.org/abs/1811.03195 [Google Scholar]
[27] Ниделл Дина, Сребро Натан и Уорд Рэйчел. 2015. Стохастический градиентный спуск, взвешенная выборка и рандомизированный алгоритм Качмарца. arXiv:1310.5715 [math.NA] [Google Scholar]
[28] Новиков Андрей. 2019.
PyClustering: библиотека интеллектуального анализа данных. Журнал программного обеспечения с открытым исходным кодом
4, 36 (апрель
2019), 1230.
[29] Pedregosa F, Varoquaux G, Gramfort A, Michel V, Thirion B, Grisel O, Blondel M, Prettenhofer P, Weiss R, Dubourg В., Вандерплас Дж., Пассос А., Курнапо Д., Бруше М., Перро М. и Дюшене Э. 2011. Scikit-learn: Машинное обучение в Python. Журнал исследований машинного обучения 12 (2011), 2825–2830. [Академия Google]
[30] Розенблатт-Розен О., Стаббингтон М.Дж.Т., Регев А. и Тейхманн С.А. 2017. Атлас клеток человека: от видения к реальности. Природа 550, 7677 (октябрь 2017), 451–453. [PubMed] [Google Scholar]
[31] Скалли Д. 2010. Web-Scale k-Means Clustering. В материалах 19-й Международной конференции по всемирной паутине (Роли, Северная Каролина, США) ( WWW’10 ). Ассоциация вычислительной техники, Нью-Йорк, штат Нью-Йорк, США, 1177–1178 гг. 10.1145/1772690.1772862 [CrossRef] [Google Scholar]
[32] Шибата Наоки и Петрогалли Франческо. 2020.
SLEEF: Портативная векторная библиотека стандартных математических функций C. Транзакции IEEE в параллельных и распределенных системах
31, 6 (июнь
2020), 1316–1327. 10.1109/tpds.2019.2960333 [CrossRef] [Google Scholar]
Транзакции IEEE в параллельных и распределенных системах
31, 6 (июнь
2020), 1316–1327. 10.1109/tpds.2019.2960333 [CrossRef] [Google Scholar]
[33] Townes FW, Hicks SC, Aryee MJ и Irizarry RA. 2019. Выбор признаков и уменьшение размеров для одноклеточной РНК-Seq на основе полиномиальной модели. Геном Биол 20, 1 (декабрь 2019), 295. [Бесплатная статья PMC] [PubMed] [Google Scholar]
[34] Ван Шибяо, Ким Джунил и Вон Гён Джэ. 2020. SHARP: сверхбыстрая и точная обработка данных секвенирования РНК одиночных клеток с помощью ансамблевой случайной проекции. Геномные исследования (2020). https://doi.org/10.1101/gr.254557.119 arXiv:https://doi.org/10.1101/gr.254557.119http://genome.cshlp.org/content/early/2020/01/28/gr. 254557.119.full.pdf+html arXiv: http://genome.cshlp.org/content/early/2020/01/28/gr.254557.119.full.pdf+html [бесплатная статья PMC] [PubMed] [Google Scholar]
[35] Вэй Юаньюань, Джанг-Жаккар Джулиан, Сабрина Фариза и Макинтош Тимоти Р. 2019 г.. MSD-Kmeans: новый алгоритм эффективного обнаружения глобальных и локальных выбросов. CoRR
абс/1910.06588 (2019). arXiv:1910.06588 http://arxiv.org/abs/1910.06588 [Google Scholar]
CoRR
абс/1910.06588 (2019). arXiv:1910.06588 http://arxiv.org/abs/1910.06588 [Google Scholar]
[36] Виттен Даниэла М.. 2011. Классификация и кластеризация данных секвенирования с использованием модели Пуассона. Анналы прикладной статистики 5, 4 (декабрь 2011), 2493–2518. 10.1214/11-aoas493 [CrossRef] [Google Scholar]
[37] Ян Вей, Билмес Джеффри и Ноубл Уильям Стаффорд. 2020. Субмодульные наброски измерений одноклеточной РНК-Seq. В материалах 11-й Международной конференции ACM по биоинформатике, вычислительной биологии и информатике здравоохранения (Виртуальное мероприятие, США) ( BCB ’20 ). Ассоциация вычислительной техники, Нью-Йорк, штат Нью-Йорк, США, статья 61, 6 страниц. 10.1145/3388440.3412409 [CrossRef] [Google Scholar]
[38] Ян Вэй, Шрайбер Джейкоб, Билмес Джеффри и Ноубл Уильям Стаффорд. 2020. Субмодульные наброски измерений одноклеточной РНК-секвенции. биоРксив (2020). https://doi.org/10.1101/2020.05.01.066738 arXiv: https://doi.org/10.1101/2020.